关于DSSP的安装教程有很多,方式也不尽相同,这里我介绍一种个人觉得最便利的方法,通过anaconda安装相关库:
conda install -c ostrokach dssp注意这种安装方式仅仅是在linux环境上才生效,关于windows下利用anaconda进行安装的方法并不是很清楚,感兴趣的可自行百度。github 上下载源码进行安装:
wget -c https://github.com/cmbi/xssp/archive/3.0.9.tar.gz
tar -xzf 3.0.9.tar.gz
cd xssp-3.0.9
./autogen.sh
./configure
make第二种方式博主没有尝试过,不能保证一定成功。
安装成功后,可以通过以下命令生成dssp文件:
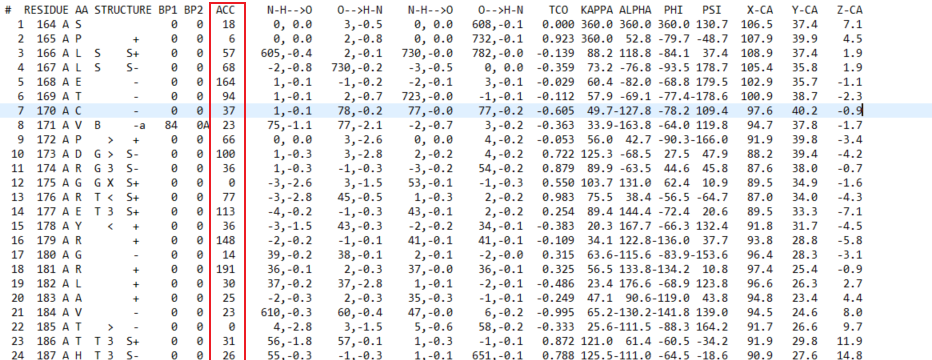
mkdssp -i 1crn.pdb -o 1crn.dssp亲测运行速度还比较快,生成的dssp文件主体信息如下图所示:
其中红框所标识出来的列就是每个残基可及表面积的数值。
当pdb文件较多时,可以使用biopython提供的dssp接口来计算dssp,下面是一个简单的脚本:
from Bio.PDB import PDBParser
from Bio.PDB.DSSP import DSSP
import numpy as np
import pandas as pd
from tqdm import tqdm
pis = pd.read_csv("pisces_no_same_id.csv")
p = PDBParser()
df = pd.DataFrame(columns=["id", "pos", "RSA"])
for index, row in tqdm(pis.iterrows()):
seq = list(row["seq"])
id_pdb = [row["id"].lower()] * len(seq)
pos = [i for i in range(1, len(seq) + 1)]
structure = p.get_structure(id_pdb[0], "pisces_pdb/{}.pdb".format(id_pdb[0]))
model = structure[0]
try:
dssp = DSSP(model, "pisces_pdb/{}.pdb".format(id_pdb[0]), dssp="../anaconda3/envs/access_project/bin/mkdssp")
except Exception as e:
print(row["id"].lower())
continue
value = dssp.property_list
rsa_value = np.array(value)[:, 3]即便是biopython提供了DSSP的API,我们仍需要安装DSSP软件,因此DSSP() 中需要指明dssp的路径,也就是使用anaconda安装dssp包的路径。
参考资料:


Good job! Thumb UP!